研究内容
[キーワード] ゲノム医科学、プレシジョン医療、がん微小環境、深層学習、数理シミュレーション、量子計算
先端の数理科学と分子観測技術で生命現象と病気を理解する
私たちの研究の目標は、がんなどの病気の克服を目指し、多くの臨床検体の生体分子の膨大なデータを深層学習などで解析することで、それらの病気に対する免疫などの生体現象の関わりを解くことです。患者さんごとに合わせた適切な種類と量の治療を施すことや、発症の予防を実現することが期待されています。それには深層学習などに基づく数理科学的な方法論を新たに考え出し、時空間的なオミクス分子データ、画像データ、臨床情報など、人の生命医科学のビッグデータを解析する必要があります。そのようにして病気の新たな原因を発見し、それらの因子間の関係を定量的なネットワークとして再構築することで、疾患メカニズムを全体のシステムとして理解します。一つの例として、がん細胞と、免疫などのがんの微小環境との潜在的な構造も含めた関係をひもとき、定量的に組み上げなおすことで、個人ごとに治療の奏効や副作用、耐性獲得などの動態を予測します。
がんとその微小環境との関係の解明
がん細胞集団と、免疫などの微小環境との関係をひもとくことで、個人ごとに治療の奏効や副作用、耐性獲得を予測します(図1-1)。例えば、私たちは肝がんの患者300人からのがん細胞の全ゲノム配列を解析し、新たな変異をもつクラスタを発見しました [1]。このクラスタの患者は、手術後にがんの再発が起こりにくく良好な経過をたどります。大腸がんでも新たなクラスタを発見しました [2]。さらに詳細に解析すると、がん細胞の集団的特性や免疫などの微小環境との関係が、クラスタ間で大きく異なることがわかってきました。がん細胞は実は私たち自身の細胞が変化したもので、非自己ですので免疫が排除するわけですが、がん細胞もまた巧みに変化して逃げます。つまり、がんの個性と、微小環境の相性や治療の内容で、その後の挙動に違いが生まれます。ほとんどのがんでは、免疫活性が高いほど予後が良いものですが、一方、免疫特権部位のがんであるブドウ膜メラノーマや低悪性度グリオーマでは、驚くべきことに、免疫活性が高いほど全生存率が悪くなることを発見し(図1-2)、そのメカニズムを解明しました [3]。このような違いをうむメカニズムをオミクスや病理画像の解析によって解明するとともに、数理シミュレーションによる治療効果予測モデルを作り、患者に合わせた治療を行うプレシジョン医療を目指します。
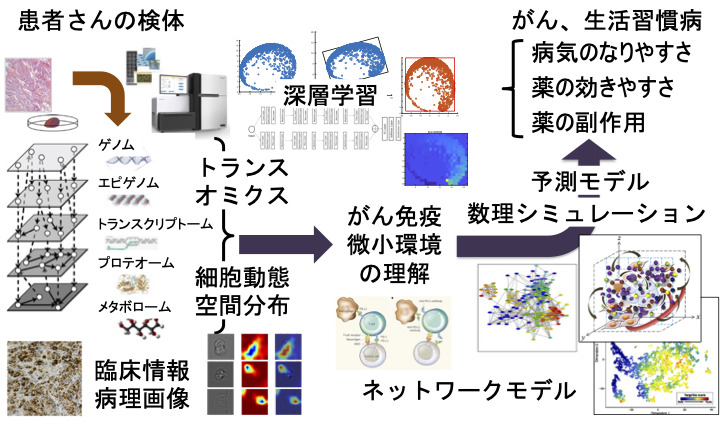
図1-1. 患者のオミクス・臨床・病理画像データで病気を解明し予測する。

図1-2. 免疫活性が高いほど全生存率が悪くなるがん種。
深層学習による独創的な解析の方法論
深層学習が、画像のみならず、ゲノムやオミクスのデータを解析する能力を極める研究をしています。一例として、オミクスデータをうまく変換して画像のように見せ深層学習にかける独自の離れ技DeepInsight法を練り出し、がんのオミクスデータでその種類を見分けられるようになりました(図2-1) [4]。さらに深層学習の内部を解析して深層学習が何を見て判別しているのかを見出す技DeepFeature法も編み出しました(図2-2) [5]。それにより、がんの個性を示す新たなシグナル伝達系を発見しました。つまり、深層学習で新たな科学的発見ができることを示せました。
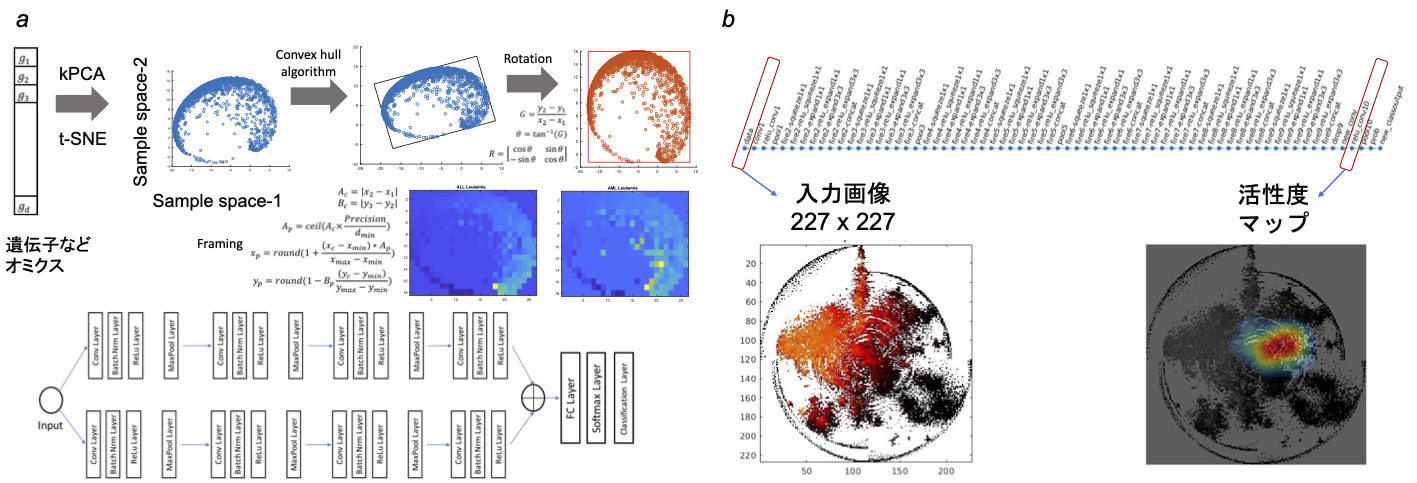
図2-1. オミクスを画像に変換して深層学習に入力する技でがんを判別 [4]。
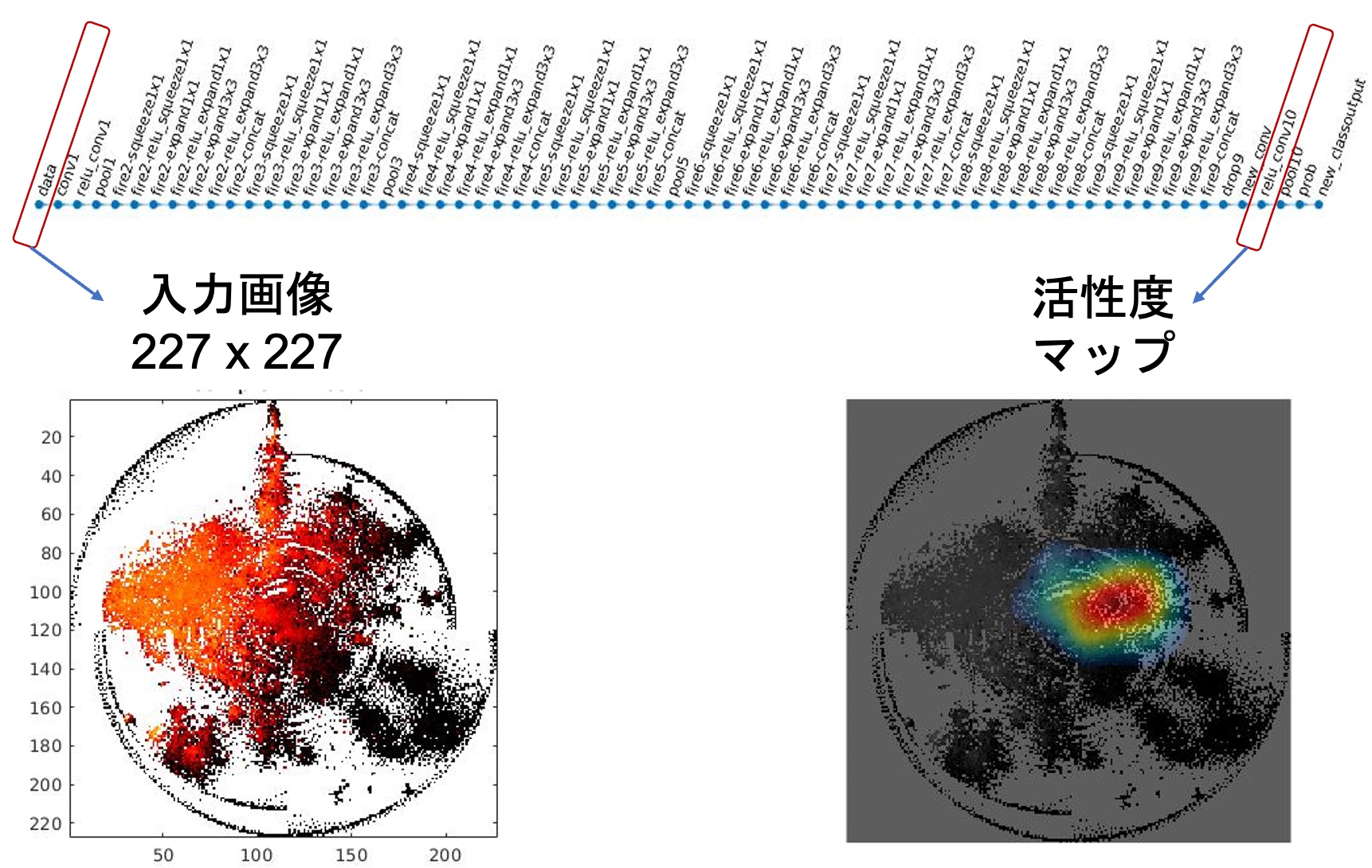
図2-2. がんを判別するとき、深層学習が何をみているかを解析する [5]。
マルチオミクスと深層学習による抗がん剤奏効予測
上記の私たちのDeepInsight法をさらに発展させ、マルチオミクスデータから患者固有の抗がん剤の奏効を予測する、深層学習を用いた新しい手法DeepInsight-3Dを提案しました(図3-1) [6]。従来の手法を大きく上回る予測性能とともに、抗がん剤の奏効の違いとなる遺伝子パターン(図3-2)と新しいパスウェイを発見しました。
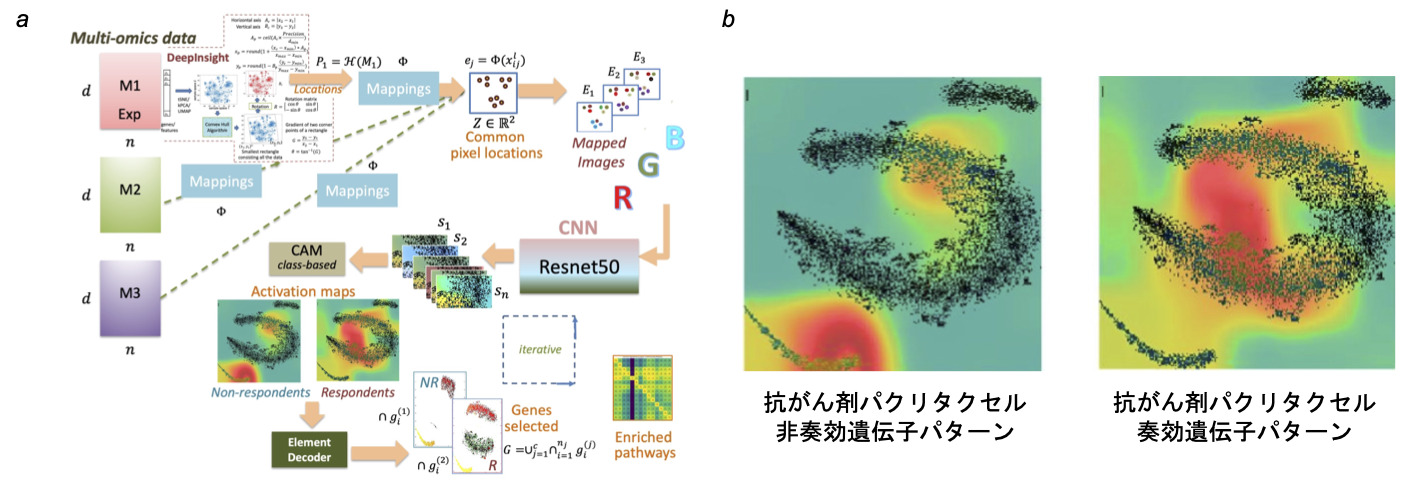
図3-1. マルチオミクスと深層学習による抗がん剤奏効予測 [6]。
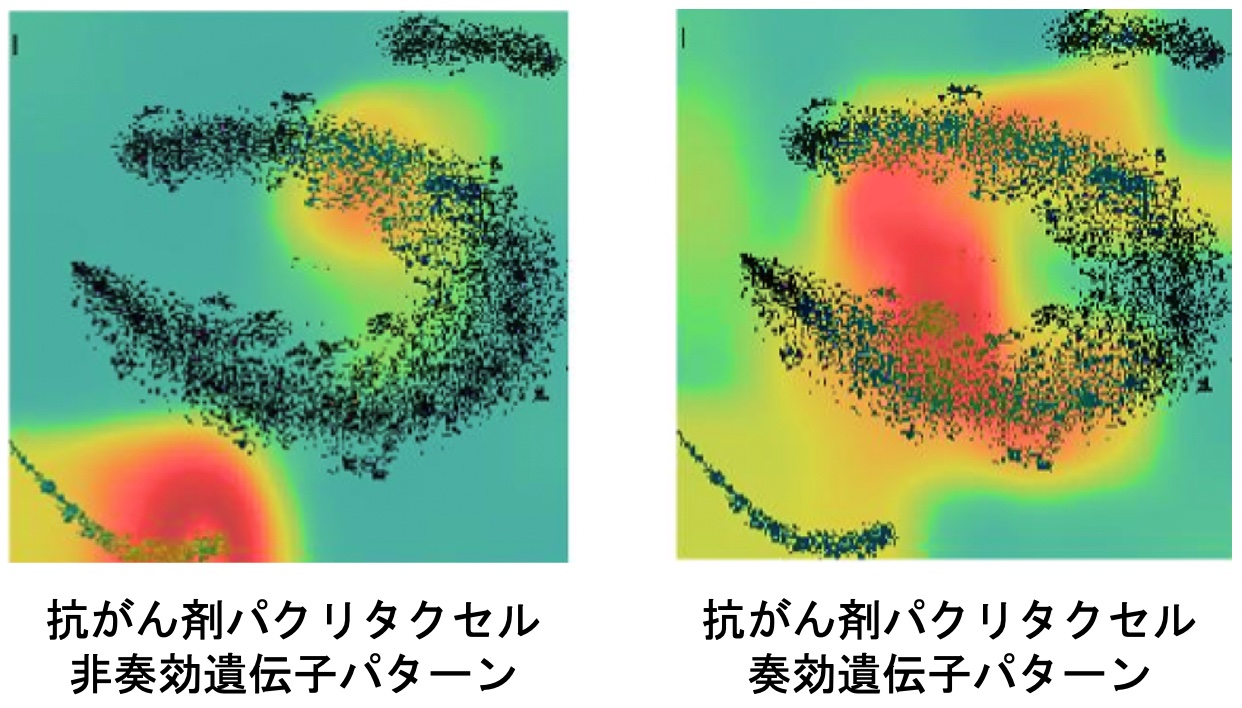
図3-2. 抗がん剤パクリタクセルへの非奏効(左)と奏効(右)に関わる遺伝子パターンの違い。
シングルセルRNA-seqデータから細胞種を同定する深層学習
上記の私たちのDeepInsight法をもとに、シングルセルRNA-seq(scRNA-seq)のデータからもとの細胞種を同定する、深層学習を用いた新しい手法scDeepInsightを提案しました(図4-1) [7]。細胞種を同定することは、scRNA-seqデータの解析によって細胞集団の不均一性を研究するための鍵となります。この手法では、scRNA-seqの非画像のデータを、私たちが独自に提案してきたDeepInsight法を使って画像に変換することにより、画像分類や特徴抽出能力が高い畳み込みニューラルネットワーク(CNN)を活用することができます。その結果、scDeepInsightは他の最先端の手法に比べてはるかに高い精度で細胞種を同定することができました(図4-2)。本提案手法により、将来的に、scRNA-seqデータを用いた生体内や疾患における細胞制御のメカニズムの解明に関わる研究に広く貢献することが期待できます。
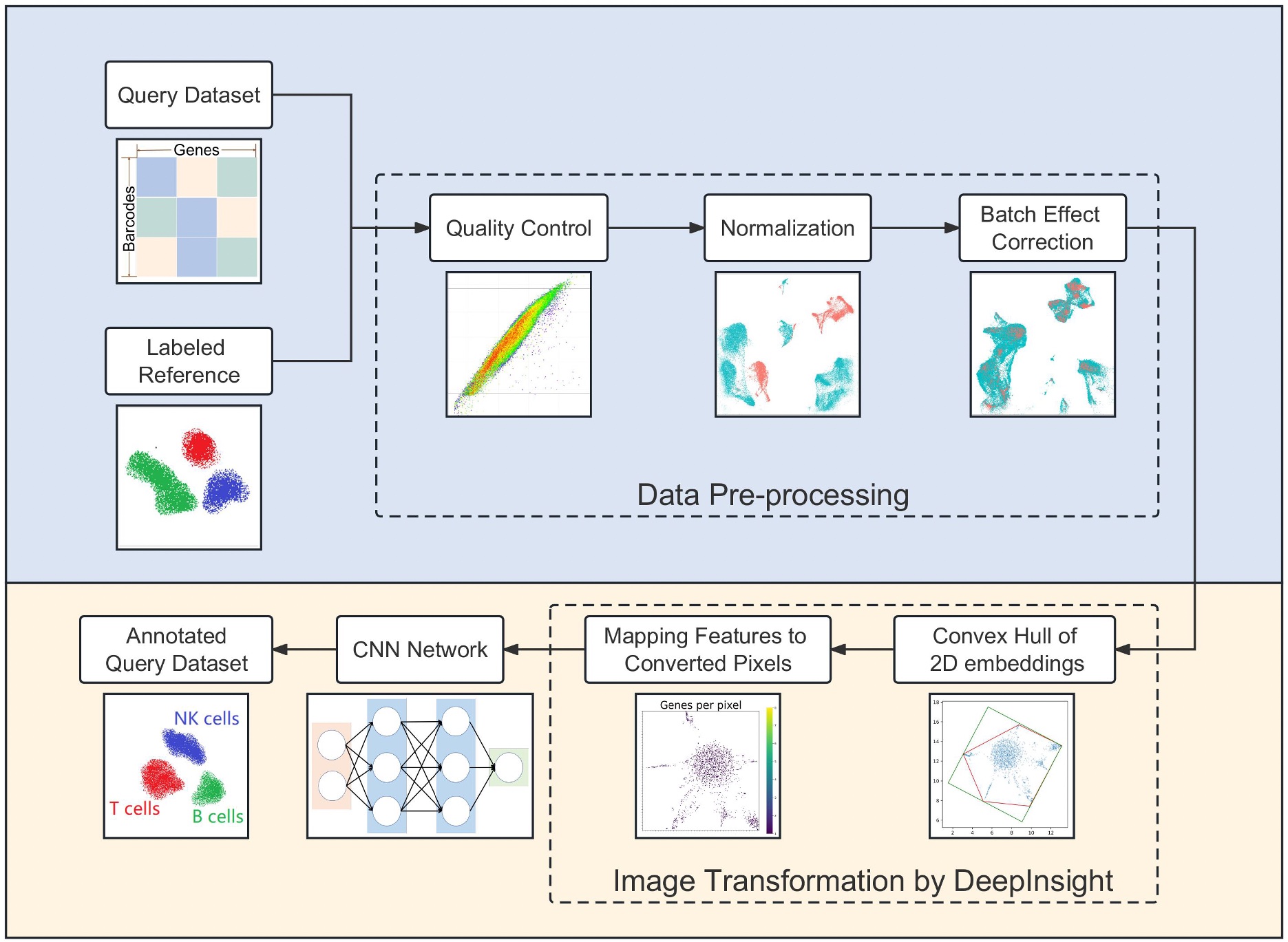
図4-1. scDeepInsightのパイプライン。
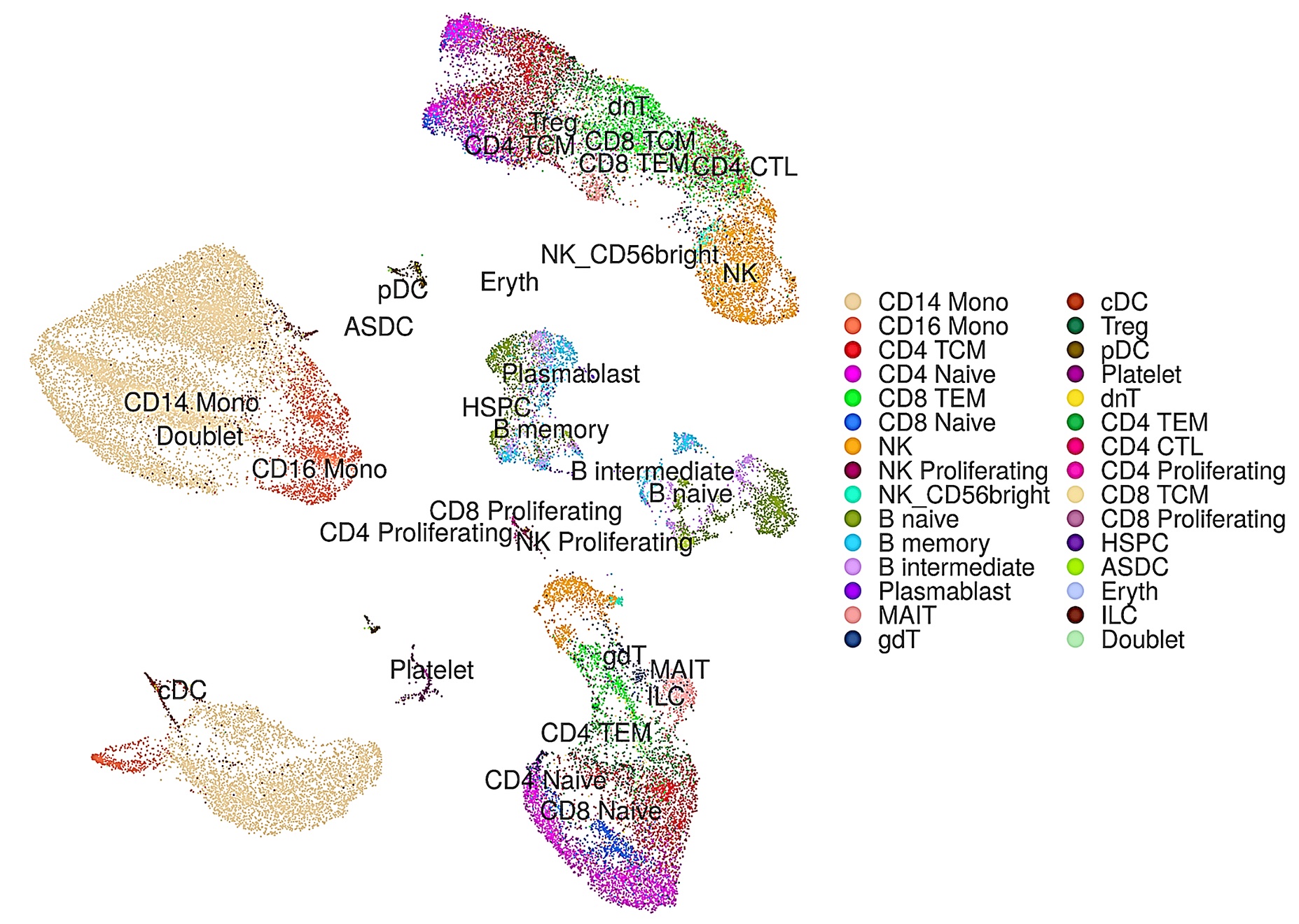
図4-2. scDeepInsightによって予測した細胞種ごとに色分けした結果。
いろいろな人が集まって研究しています
私たちの研究室は本郷キャンパスにあります。上記の研究課題に加え、量子計算機によるオミクス解析の方法の開発を行ったり、東京大学大学院理学系研究科生物科学専攻の上村研究室と連携し、独自製作したナノポアからの信号を解析して分子の識別や配列決定をする方法も開発したりしています。その他にも病院や医療機関などと多くの連携研究をしています。そのため、生命情報研究者以外にも、臨床医や、遺伝学、シークエンス解析、ネットワーク解析、数学が好きな研究者など、また、外国人研究者も多く、お互いにさまざまな考えを出しあって日々研究しています。私たちの研究室は実験よりも解析の方法論に重点を置いて医科学を推進しています。
参考文献
[1] Fujimoto A, Tsunoda T, et al. Whole genome mutational landscape and characterization of non-coding and structural mutations in liver cancer. Nature Genetics, 48, 500-509 (2016).
[2] Sugawara T, Miya F, Ishikawa T, Lysenko A, Nishino J, Kamatani T, Takemoto A, Boroevich KA, Kakimi K, Kinugasa Y, Tanabe M, Tsunoda T. Immune subtypes and neoantigen-related immune evasion in advanced colorectal cancer. iScience, 25, 1003740 (2022).
[3] Matsuo H, Kamatani T, Hamba Y, Boroevich KA, Tsunoda T. Association between high immune activity and worse prognosis in uveal melanoma and low-grade glioma in TCGA transcriptomic data. BMC Genomics, 23, 351 (2022).
[4] Sharma A, Vans E, Shigemizu D, Boroevich KA, Tsunoda T. DeepInsight: A methodology to transform a non-image data to an image for convolution neural network architecture. Scientific Reports, 9, 11399 (2019).
[5] Sharma A, Lysenko A, Boroevich KA, Vans E, Tsunoda T. DeepFeature: feature selection in nonimage data using convolutional neural network. Briefings in Bioinformatics, 22, bbab297 (2021).
[6] Sharma A, Lysenko A, Boroevich KA, Tsunoda T. DeepInsight-3D architecture for anti-cancer drug response prediction with deep-learning on multi-omics. Scientific Reports, 13, 2483 (2023).
[7] Jia S, Lysenko A, Boroevich KA, Sharma A, Tsunoda T. scDeepInsight: a supervised cell-type identification method for scRNA-seq data with deep learning. Briefings in Bioinformatics, 24, bbad266 (2023).
